Los ARN guías (también conocidos como ARNg) son un tipo particular de moléculas de ARN que guían la inserción o deleción de residuos de uridina en las moléculas de ARN mensajero mitocondrial en los protistas kinetoplastideos, en un proceso conocido como edición del ARN.
Generalidades de la edición dirigida por ARNg
Los protistas tripanosomátidos y otros kinetoplastideos poseen un novedoso proceso de modificación postranscripcional del ARN conocido como "edición del ARN". El genoma mitocondrial en estas células consiste en 20 a 50 maxicírculos que codifican para genes y criptogenes (y algunos ARNg) y entre 10 y 20 minicírculos que codifican para ARNgs. Todas estas moléculas se encuentran encadenadas en una gigantesca red de ADN que se encuentra situada en la base del flagelo dentro del compartimiento interno de la única mitocondria que estas células presentan.
La mayor parte de los transcriptos de maxicírculos no pueden ser directamente traducidos a proteínas debido a múltiples cambios de marco en sus secuencias. Estos desplazamientos de marco se corrigen luego de la transcripción por la inserción o deleción de residuos de uridina en el sitio preciso para crear un marco abierto de lectura que es traducido en proteínas mitocondriales homólogas a las proteínas mitocondriales de otras células. Las inserciones y deleciones se encuentran mediadas por unos cortos fragmentos de ARN guía (ARNgs) los cuales codifican la información de edición en forma de secuencias complementarias (permitiendo pares de bases GU como así también GC). Los ARNgs son transcriptos tanto desde los maxicírculos como desde los minicírculos.
Pasos en la edición del ARNm
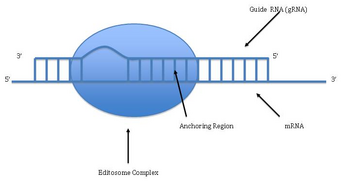
La presencia de dos genomas en las mitocondrias, uno de los cuales contiene la información de secuencias necesarias para corregir los errores en el otro genoma, es un descubrimiento reciente. El proceso de edición procede generalmente en sentido 3' a 5' sobre el ARNm. El evento inicial de edición ocurre cuando el ARNg forma un ARN dúplex con una secuencia complementaria en el ARNm un poco corriente abajo del sitio de edición. Este ARN dúplex recluta un cierto número de complejos ribonucleoproteicos que dirigen la inserción y deleción precisa de residuos de uridina, de modo tal de extender el duplex corriente arriba. El sitio de edición adyacente corriente arriba, luego es modificado de la misma manera. Una única molécula de ARNg por lo general codifica la información necesaria para varios sitios de edición (un block de edición), que producirán un dúplex ARNg/ARNm completo.
ARNm paneditados
En el caso de ARNms paneditados, el dúplex se desarma y otra molécula de ARNg se une al ARNm editado para formar un nuevo dúplex e iniciar otra ronda de edición. Las moléculas de ARNg superpuestas forman un "dominio de edición". En algunos genes hay múltiples dominios de edición. El grado de edición necesaria para un gen en particular varía entre diferentes especies de tripanosomátidos. Las variaciones consisten en la pérdida de sitios de edición en el lado 3', probablemente debido a la pérdida de secuencias provenientes de minicírculos que codifican para ARNg específicos. Se ha propuesto un modelo de retroposición para tomar cuenta de la pérdida de edición parcial y en algunos casos completa a lo largo de la evolución. La pérdida de la capacidad de edición es letal en la mayor parte de los casos, aunque se han observado pérdidas en algunas cepas de laboratorio de larga data. El mantenimiento de la capacidad de edición a lo largo de la extensa historia evolutiva de estos antiguos protistas sugiere la presencia de una ventaja selectiva, aunque la naturaleza exacta de la misma permanece aún poco clara.
Ventajas y permanencia evolutiva
Aún no resulta claro por qué los tripanosomátidos utilizan un mecanismo tan elaborado para producir ARNm. Podría haberse originado en la mitocondria temprana del ancestro del linaje de los protistas kinetoplastoideos, ya que este mecanismo se encuentra presente en los bodónidos, los cuales son ancestros de los tripanosomátidos; pero no se encuentra presente en los euglénidos, los cuales se separaron del mismo ancestro común que los kinetoplastoideos.
Ejemplo de edición, el gen MT-Cyb
En el protozoo Leishmania tarentolae, de los 18 genes mitocondriales, 12 se editan utilizando este proceso. Uno de estos genes es el Cyb.[1] Este ARNm mensajero es en realidad editado dos veces en sucesión.
En la primera edición, la secuencia relevante en el ARNm es
mRNA 5'-AAAGAAAAGGCUUUAACUUCAGGUUGU-3'
El extremo 3' se utiliza para anclar al ARNg (el ARNbg llamado gCyb-I, en este caso), por medio de un apareamiento de bases (se utilizan un algunos pares G/U). El extremo 5' no calza con precisión y una de tres endonucleasas específicas escinde al ARNm en los sitios desemparejados.
gRNA 3'-AAUAAUAAAUUUUUAAAUAUAAUAGAAAAUUGAAGUUCAGUA-5' mRNA 5'- A A AGAAA A G G C UUUAACUUCAGGUUGU-3'
A continuación el ARNm es "reparado" añadiendo sucesivas uridinas a cada sitio de edición, dando al final la secuencia:
gRNA 3' AAUAAUAAAUUUUUAAAUAUAAUAGAAAAUUGAAGUUCAGUA 5' mRNA 5' UUAUUAUUUAGAAAUUUAUGUUGUCUUUUAACUUCAGGUUGU 3'
Este gen en particular posee dos sitios de edición para ARNg superpuestos. El extremo 5' de esta sección es el punto de anclaje para otro ARNg (el gCyb-II)
Referencias
- ↑ Simpson, Larry (12 de agosto de 2009). «Uridine insertion/deletion RNA editing». UCLA. Archivado desde el original el 14 de julio de 2006. Consultado el 19 de mayo de 2006.








